Ryzyko można szacować i modelować na różne sposoby. Standardowo obliczamy wariancję stopy zwrotu i będzie to nawet poprawne, jeśli się ona nie zmienia. Pytanie tylko dlaczego uważamy, że powinna być stała? Co więcej, nawet gdy średnia wariancja się nie zmienia, to nie znaczy, że nie można znaleźć dokładniejszej miary ryzyka - taką jest wariancja warunkowa, która stanowi istotę procesu GARCH.
Pakietu rugarch w R już używałem w tym wpisie , ale tym razem skupię się tylko na nim i zrobię przykład na indeksach. Ładujemy pakiet:
if (require("rugarch")==FALSE) {
install.packages("rugarch")
}
library("rugarch")
Dodatkowo załaduję "xts", który przyda się dla prawdziwych danych oraz "forecast" do symulacji ARMA.
if (require("xts")==FALSE) {
install.packages("xts")
}
library("xts")
if (require("forecast")==FALSE) {
install.packages("forecast")
}
library("forecast")
Specyfikacja modelu
Stwórzmy sztuczny proces GARCH. Pierwsza sprawa to budowa teoretycznego modelu - specyfikacja. To się robi przy pomocy funkcji ugarchspec(). Na specyfikację składają się głównie 4 elementy:
1) model (funkcja) wariancji warunkowej: variance.model
2) model (funkcja) średniej warunkowej: mean.model
3) rozkład warunkowy zmian (np. stóp zwrotu, nie warunkowej wariancji): distribution.model
4) wartości parametrów (przy symulacji procesu należy podać wszystkie parametry modelu): fixed.pars
Ad 1) variance.model jako lista składa się głównie z dwóch części: rodzaju modelu (model) oraz rzędów (garchOrder). Z rodzajów modelu do wyboru mamy:
“sGARCH” - standardowy GARCH,
“eGARCH” - wykładniczy GARCH (The exponential GARCH)
“gjrGARCH” - Glosten-Jagannathan-Runkle GARCH
“apARCH” - The Asymmetric Power ARCH
“iGARCH” - zintegrowany GARCH (The integrated GARCH)
“csGARCH” - The Component sGARCH
“fGARCH” - full GARCH - rodzina modeli podzielona na podmodele:
a) “GARCH” - znów sGARCH
b) “TGARCH” - progowy GARCH (The Threshold GARCH)
c) “AVGARCH” - The Absolute Value GARCH
d) “NGARCH” - nieliniowy GARCH
e) “NAGARCH” - nieliniowy asymetryczny GARCH
f) “APARCH” - znowu APARCH (nie wiem dlaczego, ale to nie jest dokładnie to samo)
g) “GJRGARCH”- znowu gjrGARCH (jak wyżej)
Drugim elementem tej części są rzędy, czyli garchOrder. Należy zwrócić uwagę, że jest to dokładna analogia do ARMA(p, q), z tym że kolejność jest odwrotna: najpierw było ARCH(q), czyli średnia krocząca wariancji warunkowej, a potem dołożono część autoregresyjną tej wariancji, w sumie GARCH(q, p). Standardowo i na początek zawsze używamy q = 1, p = 1. Rzędy będą wektorem, którego elementami będzie liczba rzędów przyporządkowana danemu parametrowi. Każdy model ma swoje parametry, tak że wybierając odpowiedni typ modelu należy mieć to na względzie przy zapisywaniu tego wektora. Czyli ARCH(1) zapiszemy jako garchOrder = c(1, 0), a GARCH(1, 1) jako garchOrder = c(1, 1).
Przykład 1: ugarchspec(variance.model = list(model = "sGARCH", garchOrder = c(1, 2)))
Przykład 2, dla podmodeli: ugarchspec(variance.model = list(model = "fGARCH", submodel = "NGARCH", garchOrder = c(1, 1)))
Ad 2) mean.model jako lista zawiera dwie podstawowe części: armaOrder oraz external.regressors. Jest to po prostu ARMAX dla zmiennej, jak stopa zwrotu. Pierwszą z wymienionych części stosujemy analogicznie jak dla garchOrder. Druga część to zmienne egzogeniczne, czyli zewnętrzne regresory ARMAX. Specjalnym przypadkiem ARMAX jest GARCH-in-Mean, w którym za zmienną egzogeniczną wstawiamy samą wariancję warunkową (lub jej pierwiastek). Jest to ciekawy przykład sprzężenia zwrotnego między średnią warunkową a jej wariancją, np. między oczekiwaną (warunkową) stopą zwrotu a jej ryzykiem. Nie musimy jednak sami wyciągać tej wariancji i przyłączać do modelu, bo taki model jest do dyspozycji tutaj. Po prostu zamiast external.regressors stosujemy dodatkowy argument archm = TRUE.
Przykład1: ugarchspec(variance.model = list(model = "sGARCH", garchOrder = c(1, 1)), mean.model = list(armaOrder = c(1, 1)))
Przykład2: ugarchspec(variance.model = list(model = "sGARCH", garchOrder = c(1, 1)), mean.model = list(armaOrder = c(1, 0), archm = TRUE))
Warto dodać, że w tej części także można ustalić czy średnia ma się zachowywać zgodnie z procesem ARFIMA.
Ad 3) distribution.model, tj. rozkład prawdopodobieństwa naszej zmiennej może to być:
“norm” - rozkład normalny
“snorm” - skośno-normalny (skew-normal distribution)
“std” - t-Studenta
“sstd” skośny t-studenta (skew-student),
“ged” - uogólniony rozkład błędów (the generalized error distribution),
“sged” - skośny uogólniony rozkład błędów (the skew-generalized error distribution),
“nig” - normalny odwrotny rozkład gaussowski (the normal inverse gaussian distribution),
“ghyp” - uogólniony hiperboliczny (the Generalized Hyperbolic),
“jsu” - Johnson’s SU distribution (SU to nie skrót, ale po prostu nazwa zmiennej w tym rozkładzie, podobnie jak litera "t" w t-Studenta).
Przykład: ugarchspec(variance.model = list(model = "sGARCH", garchOrder = c(1, 2)), distribution.model = "norm")
Ad 4) fixed.pars używamy w zasadzie tylko do symulacji. Ustalamy wartości parametrów dla wybranego modelu. Nazewnictwo parametrów:
alpha1, alpha2, ... dla ARCH(q)beta1, beta2, ..., dla GARCH(p)
omega - wyraz wolny funkcji wariancji
ar1, ar2, ... dla części AR
ma1, ma2, ... dla części MA
mu dla wyrazu wolnego ARMAX
archm - wspomniany ARCH-in-Mean
arfima jako ułamkowy rząd d
mxreg1, mxreg2, ... dla zmiennych egzogenicznych (zewnętrznych regresorów) w ARMAX
Przykład 1, dla rozkładu normalnego:
ugarchspec(variance.model = list(model = "sGARCH", garchOrder = c(1, 1)),
distribution.model = "norm", fixed.pars = list(omega = 0.5,
alpha1 = 0.3,
beta1 = 0.2,
mu = 0.000001,
ar1 = 0.000001,
ma1 = 0.000001))
Przykład 2, dla skośnego rozkładu t-Studenta:
ugarchspec(variance.model = list(model = "sGARCH", garchOrder = c(1, 2)),
distribution.model = "sstd", fixed.pars = list(omega = 0.5,
alpha1 = 0.3,
beta1 = 0.2,
beta2 = 0.1,
mu = 0.000001,
ar1 = 0.000001,
ma1 = 0.000001,
skew = 1.5,
shape = 5))
Symulacja procesu GARCH
Symulację wykonamy za pomocą funkcji ugarchpath(). Głównymi argumentami są wybrany model (ze specyfikacji) oraz liczba danych. Funkcja zwraca nie tyle samą symulację, ale cały zestaw jej obiektów. Aby uzyskać same wartości, najłatwiej użyć funkcji fitted().
Przykład:
set.seed(3)
mojGarch <- ugarchspec(variance.model = list(model = "sGARCH", garchOrder = c(1, 0)),
distribution.model = "norm", fixed.pars = list(omega = 0.5,
alpha1 = 0.9,
mu = 0.000001,
ar1 = 0.000001,
ma1 = 0.000001))
symGarch <- ugarchpath(mojGarch, n.sim = 500)
symStopaZwrotu <- fitted(symGarch)
# ewentualnie
#symStopaZwrotu <- symGarch@path$seriesSim
plot(symStopaZwrotu, type="l")
To co może zaskakiwać to to, że ustawiliśmy rozkład normalny, a wykres przypomina raczej rozkład z grubymi ogonami. Jednak jest to warunkowy rozkład normalny w tym sensie, że zależy od przeszłości. Sama zmienna (stopa zwrotu) jest tu niezależna od poprzednich wartości (alpha i beta ustawione na zero), ale jej składnik losowy już tak. Składnik losowy jest po prostu procesem średniej kroczącej (normalnie nazywany MA, ale aby nie mylić z procesem dla samej zmiennej nazywany ARCH).Najlepiej połączyć obydwa wykresy w jeden: stopa zwrotu + jego zmienne odchylenie:
zakresY <- c(min(symStopaZwrotu, -symRyzyko), max(symStopaZwrotu, symRyzyko))
plot.ts(symStopaZwrotu, ylim = zakresY)
lines(symRyzyko, col = "blue")
lines(-symRyzyko, col = "blue")
Model empiryczny
Powiedzmy, że dostajemy samą taką symulację bez znajomości modelu. Chcemy dopasować otrzymane dwa szeregi do odpowiedniego modelu GARCH. Stosujemy funkcję ugarchfit(). Najważniejsze jej argumenty to dane (data), wybrany model (spec) oraz liczba prognoz poza próbą (out.sample). Zakładamy, że nic nie wiemy o próbie, więc na początek tworzymy standardowy sGARCH - oczywiście tym razem bez ustalania parametrów, bo mamy zadanie je dopiero oszacować. Za dane podstawiamy wcześniej zdefiniowaną zmienną symStopaZwrotu:
mojGarch <- ugarchspec(variance.model = list(model = "sGARCH", garchOrder = c(1, 1)), mean.model = list(armaOrder = c(1, 1)))
dopasGarch <- ugarchfit(spec = mojGarch, data = simArch, out.sample = 0)
print(dopasGarch)
*---------------------------------*
* GARCH Model Fit *
*---------------------------------*
Conditional Variance Dynamics
-----------------------------------
GARCH Model : sGARCH(1,1)
Mean Model : ARFIMA(1,0,1)
Distribution : norm
Optimal Parameters
------------------------------------
Estimate Std. Error t value Pr(>|t|)
mu 0.039416 0.040145 0.98183 0.32618
ar1 -0.427181 0.391769 -1.09039 0.27554
ma1 0.480952 0.368062 1.30671 0.19131
omega 0.598849 0.071227 8.40760 0.00000
alpha1 0.804083 0.103489 7.76977 0.00000
beta1 0.005350 0.016619 0.32191 0.74752
Robust Standard Errors:
Estimate Std. Error t value Pr(>|t|)
mu 0.039416 0.042395 0.92975 0.35250
ar1 -0.427181 0.358426 -1.19183 0.23333
ma1 0.480952 0.327567 1.46826 0.14203
omega 0.598849 0.065481 9.14534 0.00000
alpha1 0.804083 0.103111 7.79820 0.00000
beta1 0.005350 0.006337 0.84419 0.39856
LogLikelihood : -797.842
Information Criteria
------------------------------------
Akaike 3.2154
Bayes 3.2659
Shibata 3.2151
Hannan-Quinn 3.2352
Weighted Ljung-Box Test on Standardized Residuals
------------------------------------
statistic p-value
Lag[1] 1.067 0.3016
Lag[2*(p+q)+(p+q)-1][5] 1.357 0.9994
Lag[4*(p+q)+(p+q)-1][9] 3.017 0.8880
d.o.f=2
H0 : No serial correlation
Weighted Ljung-Box Test on Standardized Squared Residuals
------------------------------------
statistic p-value
Lag[1] 0.02626 0.8713
Lag[2*(p+q)+(p+q)-1][5] 0.21540 0.9914
Lag[4*(p+q)+(p+q)-1][9] 1.84882 0.9223
d.o.f=2
Weighted ARCH LM Tests
------------------------------------
Statistic Shape Scale P-Value
ARCH Lag[3] 0.001223 0.500 2.000 0.9721
ARCH Lag[5] 0.274535 1.440 1.667 0.9470
ARCH Lag[7] 1.653454 2.315 1.543 0.7901
Nyblom stability test
------------------------------------
Joint Statistic: 1.0574
Individual Statistics:
mu 0.03849
ar1 0.33674
ma1 0.31603
omega 0.38146
alpha1 0.21883
beta1 0.27590
Asymptotic Critical Values (10% 5% 1%)
Joint Statistic: 1.49 1.68 2.12
Individual Statistic: 0.35 0.47 0.75
Sign Bias Test
------------------------------------
t-value prob sig
Sign Bias 0.9554 0.3399
Negative Sign Bias 0.1454 0.8845
Positive Sign Bias 0.2359 0.8136
Joint Effect 1.2101 0.7506
Adjusted Pearson Goodness-of-Fit Test:
------------------------------------
group statistic p-value(g-1)
1 20 16.00 0.6573
2 30 28.96 0.4672
3 40 34.40 0.6796
4 50 53.60 0.3023Wydruk dostarcza nie tylko estymacji współczynników, ale też wszystkich potrzebnych informacji o poprawności modelu. Wykonujemy jego diagnostykę.
Są dwie wersje testowania istotności parametrów. Pierwsza to konwencjonalna metoda największej wiarygodności (Optimal parameters), a druga odporna na nieprawidłowe założenie rozkładu (Robust Standard Errors). Ta druga jest więc mocniejsza i powinniśmy na nią patrzeć jako pierwszą. Parametry się w niej nie zmieniają, a jedynie ich przedziały istotności. Widzimy, że jedynie istotne są omega i alpha1, czyli poprawnie, natomiast same ich wartości też dość dobrze oszacowane (alpha1 jest równy 0.9, a oszacowano go na 0.8; omega równa się 0.5, a w estymacji = 0.6). Oczywiście widząc, że beta1 oraz ma1 są nieistotne, moglibyśmy poprawić teraz model. Dokończmy jednak najpierw diagnostykę.
Dalej mamy kryteria informacyjne (Information Criteria), które w tym wypadku nic nam nie powiedzą, bo nie porównujemy modelu z innym.
Ważony test Ljunga-Boxa sprawdzamy na dwóch poziomach: resztach (Weighted Ljung-Box Test on Standardized Residuals) oraz kwadratach reszt (Weighted Ljung-Box Test on Standardized Squared Residuals). Jeśli p-value > 0.05, to możemy przyjąć, że nie występują autokorelacje w pierwszym przypadku w resztach, w drugim - w kwadratach reszt. Ponieważ p-value dla wielu rzędów q i p jest mocno powyżej 0.05, przyjęty model jest poprawny.
Podobnym testem jest Weighted ARCH LM, ale skupia się na samym efekcie ARCH, a dokładniej czy przyjęty model nie pominął tego efektu, np. dla pewnego rzędu. Jeżeli p-value > 0.05, to znaczy, że nie pominął, w przeciwnym wypadku prawdopodobnie pominął. Widzimy, że dla wielu rzędów q i p jest mocno powyżej 0.05, przyjęty model jest poprawny.
Test Nybloma (Nyblom stability test) bada stabilność modelu, tzn. czy parametry zmieniają się w czasie. Jest to bardzo ważny test z punktu widzenia właśnie analizy finansowej. Joint Statistic: 1.0574 dotyczy stabilności całego modelu. Porównujemy to z teoretycznymi wartościami dla kolejnych p-value, szczególnie patrzymy na wartość korespondującą z 5% - tutaj jest to 1.68. Ponieważ 1.68 > 1.0574 , nie ma podstaw do odrzucenia hipotezy zerowej o stabilności modelu. Dodatkowa analiza dotyczy każdego współczynnika osobno. Patrzymy na Individual Statistic. Mamy tu współczynniki:
mu = 0.03849, ar1 = 0.33674, ma1 = 0.31603, omega 0.38146, alpha1 0.21883, beta1 0.27590
Wartości te porównujemy z wartością teoretyczną odpowiadającą 5% - wynosi 0.47. Ponieważ 0.47 jest większa od każdego wspołczynnika, to znaczy, że wszystkie parametry są stabilne, zarówno średniej, wariancji, jak i rozkładu.
Test błędu znaku (Sign Bias Test) pozwala z kolei ocenić czy w szeregu nie występują szoki dodatnie lub ujemne. Niekonsekwentne oznaczenie prob sig to nic innego jak p-value. Zarówno dla dodatniego, ujemnego, jak i łącznego efektu, jest ono większe od 0.05, czyli szoki nie występują. Gdyby występowały, trzeba by zmienić model na EGARCH, APARCH, TGARCH lub gjrGARCH.
W końcu test dobroci dopasowania modelu (Adjusted Pearson Goodness-of-Fit Test) sprawdza czy rozkład reszt jest odpowiedni. Mamy tu podział na kolejne kwantyle, tak że możemy zobaczyć jak reszty się zachowują w całym rozkładzie. Jeśli p-value > 0.05 dla któregoś kwantyla, znaczyłoby, że przyjęty rozkład jest niewłaściwy. W tym wypadku wszystkie wskazują poprawność rozkładu - przyjęliśmy normalny i oceniony jako też normalny.
Diagnostyka zakończona. Tutaj warto wspomnieć ciekawą opcję, którą można by nazwać graficzną diagnostyką. Wystarczy wpisać:
plot(dopasGarch)
i dostaniemy listę:
Make a plot selection (or 0 to exit):
1: Series with 2 Conditional SD Superimposed
2: Series with 1% VaR Limits
3: Conditional SD (vs |returns|)
4: ACF of Observations
5: ACF of Squared Observations
6: ACF of Absolute Observations
7: Cross Correlation
8: Empirical Density of Standardized Residuals
9: QQ-Plot of Standardized Residuals
10: ACF of Standardized Residuals
11: ACF of Squared Standardized Residuals
12: News-Impact Curve
| |
|
Wybierzmy 8:
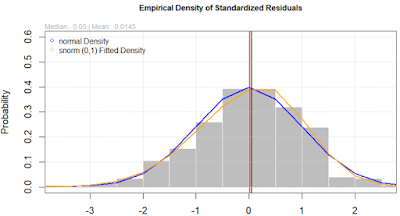
Dzięki tej opcji możemy naocznie ocenić poprawność przyjętego rozkładu.
Również zamiast testu dobroci możemy użyć wykresu kwantylowego (QQ), tzn. wybieramy z listy 9:
Tak jak wspomniałem, skoro niektóre parametry okazały się nieistotne, to moglibyśmy teraz model poprawić, tak że kod wyglądałby następująco:
mojGarch <- ugarchspec(variance.model = list(model = "sGARCH", garchOrder = c(1, 0)), mean.model = list(armaOrder = c(0, 0)))
print(dopasGarch)
Poniżej wydruk tylko kilku pierwszych statystyk:
*---------------------------------*
* GARCH Model Fit *
*---------------------------------*
Conditional Variance Dynamics
-----------------------------------
GARCH Model : sGARCH(1,0)
Mean Model : ARFIMA(0,0,0)
Distribution : norm
Optimal Parameters
------------------------------------
Estimate Std. Error t value Pr(>|t|)
mu 0.039045 0.039600 0.98599 0.32414
omega 0.617144 0.067416 9.15423 0.00000
alpha1 0.800232 0.102546 7.80364 0.00000
Robust Standard Errors:
Estimate Std. Error t value Pr(>|t|)
mu 0.039045 0.042187 0.92552 0.35469
omega 0.617144 0.063213 9.76291 0.00000
alpha1 0.800232 0.102620 7.79800 0.00000
LogLikelihood : -799.3085
Information Criteria
------------------------------------
Akaike 3.2092
Bayes 3.2345
Shibata 3.2092
Hannan-Quinn 3.2192Chociaż wiemy już, że drugi model jest lepszy, to aby ocenić to obiektywnie, porównamy ich kryteria informacyjne, które właśnie tutaj się przydadzą. Jeżeli wszystkie kryteria: AIC, BIC, SIC i HQIC wskazują mniejsze wartości, to znaczy, że ten model jest lepszy. Tak rzeczywiście się dzieje
Akaike 3.2092 < 3.2154Bayes 3.2345 < 3.2659Shibata 3.2092 < 3.2151Hannan-Quinn 3.2192 < 3.2352Otrzymujemy jednoznaczny wniosek, że ten drugi model jest lepszy od pierwszego.
Prognoza
Mając dobry model możemy przejść do prognozy. Prognozę można wykonać zarówno dla stopy zwrotu, jak i jej ryzyka. Ale ponieważ prognoza stopy zwrotu = oczekiwana stopa zwrotu, to znaczy, że w naszym przypadku jest to po prostu wyraz wolny bliski zera (nie mamy tu procesu ARMA), więc tak naprawdę interesuje nas tylko prognoza ryzyka. Prognozę wykonujemy przy pomocy ugarchforecast(). Jej główne argumenty to
1. model empiryczny z ugarchfit, ewentualnie model teoretyczny z podaniem danych (fitORspec)
2. ile wykonać prognoz do przodu (n.ahead)
3. ile prognoz ma się nakładać, aby stworzyć pierwszą poza próbą (n.roll)
4. ile ostatnich danych ma być zachowanych poza testowaną próbą (out.sample)
Punkty 3 i 4 są powiązane i na razie je zostawiam.
Stworzymy prognozę dla pięciu nowych punktów:
noweOkresy <- 5
prognozaGarch <- ugarchforecast(dopasGarch, n.ahead = noweOkresy)
prognozaStopaZwrotu <- fitted(prognozaGarch)
prognozaRyzyko <- sigma(prognozaGarch)
Aby zobaczyć to wszystko graficznie, powinniśmy połączyć próbę z prognozą. Jednak łączenie różnych typów danych i pokazywanie na wykresie może być w R wyzwaniem. Ponieważ działamy na szeregach czasowych, to najpierw zamieńmy wszystkie dane na ts i połączymy z prognozą:
# zamiana na ts i łączenie z prognozą
stopaZwrotu <- as.ts(symStopaZwrotu)
ryzyko <- as.ts(symRyzyko)
ostatniOkres <- end(stopaZwrotu)
dlugoscProby <- length(stopaZwrotu)
stopaZwrotu <- ts(c(as.numeric(stopaZwrotu), rep(NA, noweOkresy)), start = start(stopaZwrotu))
prognozaStopaZwrotu <- ts(c(stopaZwrotu[dlugoscProby], as.numeric(prognozaStopaZwrotu)), start = ostatniOkres)
ryzyko <- ts(c(symRyzyko, rep(NA, noweOkresy)), start = start(stopaZwrotu))
prognozaRyzyko <- ts(c(ryzyko[dlugoscProby], as.numeric(prognozaRyzyko)), start = ostatniOkres)
Pokażmy 30 ostatnich obserwacji i 5 nowych
# wykres
zakresX <- c(dlugoscProby - 30, length(stopaZwrotu))
zakresY <- c(min(na.omit(stopaZwrotu), -na.omit(ryzyko)), max(na.omit(stopaZwrotu), -na.omit(ryzyko)) + 2)
plot(stopaZwrotu, xlim=zakresX, ylim=zakresY, lwd=2)
lines(prognozaStopaZwrotu, lwd=2, col="green")
lines(ryzyko, lwd=2, col="red")
lines(-ryzyko, lwd=2, col="red")
lines(prognozaRyzyko, lwd=2, col="brown")
lines(-prognozaRyzyko, lwd=2, col="brown")
legend(x="topleft", legend=c("stopa zwrotu", "prognoza stopy zwrotu", "modelowane ryzyko",
"prognoza ryzyka"), lwd=c(2,2,2,2), col=c("black",
"green",
"red",
"brown"))
Ostatnia symulacja testowa
Na koniec przeprowadzę ostatnią symulację i jej test. Tym razem na odwrót - stworzę zwykły proces AR(1) o 500 obserwacjach i sprawdzę jak rugarch radzi sobie z tym, gdy ma zły model.
set.seed(3)
symStopaZwrotu = arima.sim(list(order=c(1,0,0), ar=0.6), n=500, mean=1)
mojGarch <- ugarchspec(variance.model = list(model = "sGARCH",
garchOrder = c(1, 1)),
mean.model = list(armaOrder = c(1, 0)))
dopasGarch <- ugarchfit(spec = mojGarch, data = symStopaZwrotu, out.sample = 0)
print(dopasGarch)
Częściowy wydruk:
Robust Standard Errors:
Estimate Std. Error t value Pr(>|t|)
mu 2.607355 0.115327 22.608 0.00000
ar1 0.605802 0.033249 18.220 0.00000
omega 0.001275 0.003960 0.322 0.74745
alpha1 0.000000 0.003644 0.000 1.00000
beta1 0.999000 0.000065 15463.568 0.00000
LogLikelihood : -724.7
Information Criteria
------------------------------------
Akaike 2.9188
Bayes 2.9610
Shibata 2.9186
Hannan-Quinn 2.9354O ile algorytm prawidłowo oszacował ar1 na 0.6 i alpha1 na zero, o tyle błędnie przypisał becie 1. Ta pełna jedynka pokazuje, że nie możemy kierować się bezmyślnie samą istotnością: skoro beta równa się 1, to może znaczyć, że model został źle wyspecyfikowany. Aby to ocenić, usuńmy betę ze specyfikacji:
mojGarch <- ugarchspec(variance.model = list(model = "sGARCH",
garchOrder = c(1, 0)),
mean.model = list(armaOrder = c(1, 0)))
dopasGarch <- ugarchfit(spec = mojGarch, data = symStopaZwrotu, out.sample = 0)
print(dopasGarch)
Robust Standard Errors:
Estimate Std. Error t value Pr(>|t|)
mu 2.60768 0.120767 21.593 0
ar1 0.60566 0.035427 17.096 0
omega 1.06510 0.081482 13.072 0
alpha1 0.00000 0.048245 0.000 1
LogLikelihood : -725
Information Criteria
------------------------------------
Akaike 2.9160
Bayes 2.9497
Shibata 2.9158
Hannan-Quinn 2.9292Teraz widzimy, że model rzeczywiście był źle wyspecyfikowany, ponieważ wszystkie kryteria się poprawiły (mają mniejszą wartość). Jedynie omega zmieniła wartość, a więc to ją beta1 wcześniej psuła. Oczywiście w symulacji nie ustawialiśmy odchylenia standardowego, a więc zostało ono automatycznie ustalone na 1, a wobec tego jego kwadrat - omega - też musi się równać 1 i tyle też wyniosła estymacja (i również w końcu stała się istotna). Jedynym błędem jaki się nam ostał jest zbyt duży wyraz wolny (miał się równać 1). Ten błąd jednak nie wynika z błędnego algorytmu, bo gdy będziemy estymować zwykłą arimą:
arima(x=symStopaZwrotu, order=c(1,0,0))
dostaniemy to samo. Dopiero użycie MNK:
summary(lm(symStopaZwrotu[-1] ~ symStopaZwrotu[-length(symStopaZwrotu)]))
przyniesie prawidłowy wyraz wolny równy ok. 1. Na marginesie, w tej próbce akurat średnia arytmetyczna rzeczywiście równa się 2.6, co wynika po prostu z tego, że składa się ze średniej niewarunkowej i warunkowej. MNK lepiej oddziela te dwie części. Algorytmy dla ARIMA i GARCH są przeznaczone dla bardziej złożonych modeli, a nie zwykłego AR, stąd kosztem są takie błędy.
Model na prawdziwych danych
Dzienne stopy zwrotu WIG20
Zakres czasu: 2023-04-21 - 2024-07-02, czyli ostatnie 300 sesji. Dane ściągnięte ze stooq.pl. Przygotowujemy analizę:
# ustawiamy folder roboczy
sciezka = r"(C:\testy)"
#zamieniam "\" na "/"
sciezka = gsub("\\", "/", sciezka, fixed=T)
setwd(sciezka_testy)
nazwaPliku = "wig20_d.csv"
plik <- read.csv(nazwaPliku)
cena <- plik$Zamkniecie
Obs <- 300
stopaZwrotu <- tail(diff(log(cena)), Obs)
okres <- as.Date(tail(plik$Data[-1], Obs))
Zwracam uwagę, że używam logarytmicznej stopy zwrotu, co generalnie ułatwia nie tylko kodowanie, ale testowanie (zmienna staje się "bardziej" normalna). Ostatnie 300 stóp zwrotu WIG20 okazały się trudniejsze do modelowania niż sądziłem. Przykładowo, ten sam model co dla symulacji, okazał się niedobry, ponieważ parametr ARCH(1) był nieistotny:
mojGarch <- ugarchspec(variance.model = list(model = "sGARCH", garchOrder = c(1, 1)), mean.model = list(armaOrder = c(1, 1)))
dopasGarch <- ugarchfit(spec = mojGarch, data = simArch, out.sample = 0)
print(dopasGarch)
*---------------------------------*
* GARCH Model Fit *
*---------------------------------*
Conditional Variance Dynamics
-----------------------------------
GARCH Model : sGARCH(1,0)
Mean Model : ARFIMA(0,0,0)
Distribution : norm
Optimal Parameters
------------------------------------
Estimate Std. Error t value Pr(>|t|)
mu 0.000933 0.000713 1.3079 0.19092
omega 0.000153 0.000014 11.0540 0.00000
alpha1 0.000000 0.039697 0.0000 1.00000
Robust Standard Errors:
Estimate Std. Error t value Pr(>|t|)
mu 0.000933 0.000661 1.4103 0.15844
omega 0.000153 0.000016 9.3305 0.00000
alpha1 0.000000 0.033403 0.0000 1.00000
LogLikelihood : 892.5
Information Criteria
------------------------------------
Akaike -5.9298
Bayes -5.8928
Shibata -5.9300
Hannan-Quinn -5.9150
Zobaczmy z jak ogromną kombinacją parametrów do określenia mamy tu do czynienia: rodzaj modelu zarówno GARCH, jak i ARFIMA (w tym określenie czy GARCH umieścić w ARMA czy nie) , rzędy GARCH(q, p), rząd AR, rząd MA, typ rozkładu. Wykonałem wiele testów i doszedłem (przynajmniej na dziś) do wniosku, że najlepszym modelem jest TGARCH(1, 1)-in-Mean z rozkładem skośno-normalnym. TGARCH uwzględnia inne zachowanie się warunkowej wariancji, gdy są spadki i inne, gdy są wzrosty. Dostajemy więc dużo dokładniejsze zmiany ryzyka w kolejnych okresach niż w standardowym GARCH. Nazwa TGARCH pochodzi od Threshold GARCH, czyli progowy GARCH. Progiem jest tu... zero, poniżej którego następuje przeskoczenie na parametr dla ujemnych zmian (nie jest to nic skomplikowanego, po prostu rozdziela się składnik losowy na część dodatnią i ujemną).
mojGarch <- ugarchspec(variance.model = list(model = "fGARCH", submodel = "TGARCH",
garchOrder = c(1, 1)),
mean.model = list(armaOrder = c(0, 0), archm = T), distribution.model = "snorm")
dopasGarch <- ugarchfit(spec = mojGarch, data = stopaZwrotu, out.sample = 0)
print(dopasGarch)
*---------------------------------*
* GARCH Model Fit *
*---------------------------------*
Conditional Variance Dynamics
-----------------------------------
GARCH Model : fGARCH(1,1)
fGARCH Sub-Model : TGARCH
Mean Model : ARFIMA(0,0,0)
Distribution : snorm
Optimal Parameters
------------------------------------
Estimate Std. Error t value Pr(>|t|)
mu -0.011509 0.000678 -16.983 0
archm 1.029024 0.057292 17.961 0
omega 0.000539 0.000038 14.082 0
alpha1 0.041037 0.003865 10.618 0
beta1 0.922511 0.000036 25297.879 0
eta11 1.000000 0.182817 5.470 0
skew 1.161197 0.095820 12.118 0
Robust Standard Errors:
Estimate Std. Error t value Pr(>|t|)
mu -0.011509 0.000354 -32.488 0
archm 1.029024 0.023033 44.677 0
omega 0.000539 0.000018 30.293 0
alpha1 0.041037 0.001523 26.947 0
beta1 0.922511 0.000050 18517.286 0
eta11 1.000000 0.075490 13.247 0
skew 1.161197 0.089539 12.969 0
LogLikelihood : 902.5
Information Criteria
------------------------------------
Akaike -5.9702
Bayes -5.8838
Shibata -5.9713
Hannan-Quinn -5.9356
Weighted Ljung-Box Test on Standardized Residuals
------------------------------------
statistic p-value
Lag[1] 0.1093 0.7409
Lag[2*(p+q)+(p+q)-1][2] 0.5386 0.6758
Lag[4*(p+q)+(p+q)-1][5] 1.9115 0.6395
d.o.f=0
H0 : No serial correlation
Weighted Ljung-Box Test on Standardized Squared Residuals
------------------------------------
statistic p-value
Lag[1] 1.715 0.1903
Lag[2*(p+q)+(p+q)-1][5] 2.352 0.5379
Lag[4*(p+q)+(p+q)-1][9] 3.638 0.6506
d.o.f=2
Weighted ARCH LM Tests
------------------------------------
Statistic Shape Scale P-Value
ARCH Lag[3] 0.7304 0.500 2.000 0.3928
ARCH Lag[5] 1.2072 1.440 1.667 0.6726
ARCH Lag[7] 2.2430 2.315 1.543 0.6659
Nyblom stability test
------------------------------------
Joint Statistic: 1.287
Individual Statistics:
mu 0.11514
archm 0.10348
omega 0.08008
alpha1 0.06191
beta1 0.06585
eta11 0.08665
skew 0.19526
Asymptotic Critical Values (10% 5% 1%)
Joint Statistic: 1.69 1.9 2.35
Individual Statistic: 0.35 0.47 0.75
Sign Bias Test
------------------------------------
t-value prob sig
Sign Bias 0.3681 0.7131
Negative Sign Bias 1.3763 0.1698
Positive Sign Bias 1.1213 0.2631
Joint Effect 3.1698 0.3662
Adjusted Pearson Goodness-of-Fit Test:
------------------------------------
group statistic p-value(g-1)
1 20 14.67 0.7435
2 30 26.80 0.5825
3 40 31.47 0.7990
4 50 44.00 0.6756Na pierwszy rzut oka może się wydać dziwne, że niewarunkowa średnia stopa zwrotu jest ujemna (mu = -0.0115), ale trzeba pamiętać, że model zawiera dodatkowy element - średnią warunkową, która zależy od warunkowego odchylenia standardowego i jej parametr jest dodatni. Powoduje to, że średnia stopa zwrotu zmienia się w czasie. Warto odnotować, że taki efekt może wcale nie zostać wykryty przez testy stabilności.
Aby zobaczyć czy rozkład się zgadza, można użyć komendy
plot(dopasGarch, which=8)
i dodatkowo
plot(dopasGarch, which=9)
Wykonujemy prognozę. Zauważmy, że w tym przypadku możemy nieco poprawić precyzję prognozowanej stopy zwrotu: nie jest to po prostu wyraz wolny (średnia niewarunkowa), ale funkcja ryzyka (wariancji warunkowej). Z tego powodu wyłuskamy z empirycznego modelu nie tylko estymację ryzyka z próby, ale też "prognozy" (oczekiwanej stopy zwrotu) z próby. Prognozy poza próbą na 1 dzień do przodu także zrobimy.
# prognoza w próbie (dopasowanie do próby)
stopaZwrotuDopas <- fitted(dopasGarch)
ryzyko <- sigma(dopasGarch)
# prognoza poza próbą
ileDoPrzodu <- 1
prognozaGarch <- ugarchforecast(dopasGarch, n.ahead = ileDoPrzodu)
prognozaStopaZwrotu <- fitted(prognozaGarch)
prognozaRyzyko <- sigma(prognozaGarch)
# zamiana na ts i łączenie z prognozą dla wykresu
stopaZwrotuWykres <- as.ts(stopaZwrotu)
stopaZwrotuDopasWykres <- as.ts(stopaZwrotuDopas)
ryzykoWykres <- as.ts(ryzyko)
ostatniOkres <- end(stopaZwrotuWykres)
dlugoscProby <- length(stopaZwrotuWykres)
stopaZwrotuWykres <- ts(c(as.numeric(stopaZwrotuWykres), rep(NA, ileDoPrzodu)), start = start(stopaZwrotuWykres))
stopaZwrotuDopasWykres <- ts(c(stopaZwrotuDopasWykres, rep(NA, ileDoPrzodu)), start = start(stopaZwrotuWykres))
prognozaStopaZwrotu <- ts(c(rep(NA, dlugoscProby-1), stopaZwrotuDopasWykres[dlugoscProby],
as.numeric(prognozaStopaZwrotu)), start = stopaZwrotuWykres)
ryzykoWykres <- ts(c(ryzykoWykres, rep(NA, ileDoPrzodu)), start = start(stopaZwrotuWykres))
prognozaRyzyko <- ts(c(rep(NA, dlugoscProby-1), ryzykoWykres[dlugoscProby], as.numeric(prognozaRyzyko)), start = stopaZwrotuWykres)
okres <- tail(plik$Data[-1], Obs)
okresPrognoza <- max(okres) + days(1)
okresWykres <- c(okres, okresPrognoza)
# wykres
Ost <- 15
zakresX <- c(okresWykres[dlugoscProby - Ost + 1], okresWykres[length(okresWykres)])
zakresY <- c(min(na.omit(stopaZwrotuWykres), -na.omit(ryzykoWykres)), max(na.omit(stopaZwrotuWykres), -na.omit(ryzykoWykres)) + max(na.omit(stopaZwrotuWykres), -na.omit(ryzykoWykres)))
plot(x=okresWykres, y=stopaZwrotuWykres, main="Dzienna stopa zwrotu WIG20", xaxt="n", xlim=zakresX, ylim=zakresY, lwd=2, type="l", xlab="")
axis(side=1, at=okresWykres, labels=okresWykres, las=2, cex.axis=0.8)
lines(x=okresWykres, y=stopaZwrotuDopasWykres, col="lightblue", lwd=2)
lines(x=okresWykres, y=prognozaStopaZwrotu, lwd=2, col="green")
lines(x=okresWykres, y=ryzykoWykres, lwd=2, col="red")
lines(x=okresWykres, y=-ryzykoWykres, lwd=2, col="red")
lines(x=okresWykres, y=prognozaRyzyko, lwd=2, col="brown")
lines(x=okresWykres, y=-prognozaRyzyko, lwd=2, col="brown")
abline(h=0, col="grey")
legend(x="topleft", legend=c("stopa zwrotu", "modelowana stopa zwrotu (GARCH-In-Mean)", "prognoza stopy zwrotu", "modelowane ryzyko (T-GARCH)",
"prognoza ryzyka (T-GARCH)"), lwd=c(2,2,2,2,2), col=c("black",
"lightblue",
"green",
"red",
"brown"))
Miesięczne stopy zwrotu
Aby zagregować dane dzienne do miesięcznych użyłem pakietu xts. Obserwacji jest tak samo 300, więc cofamy się znacznie dalej w przeszłość. Zakres to lipiec 1999 - czerwiec 2024.
tabela <- as.data.frame(to.monthly(as.xts(plik), indexAt="endof", OHLC=FALSE))
cena <- tabela$Zamkniecie
Obs <- 300
stopaZwrotu <- tail(diff(log(cena)), Obs)
okres <- as.Date(tail(rownames(tabela)[-1], Obs))
Tym razem lepsze rezultaty daje EGARCH:
mojGarch <- ugarchspec(variance.model = list(model = "eGARCH", submodel = NULL,
garchOrder = c(1, 1)),
mean.model = list(armaOrder = c(0, 0), archm = T), distribution.model = "snorm")
I dalej już analogicznie do dziennych. Jedyną różnicą w kodzie będzie zastąpienie funkcji days(1) przez month(1).
okresPrognoza <- max(okres) + days(1)
Częściowy wydruk:
Robust Standard Errors:
Estimate Std. Error t value Pr(>|t|)
mu -0.046871 0.002481 -18.8899 0.000000
archm 0.740374 0.038708 19.1271 0.000000
omega -0.340842 0.042694 -7.9834 0.000000
alpha1 -0.081079 0.021606 -3.7526 0.000175
beta1 0.938421 0.007192 130.4857 0.000000
gamma1 0.129712 0.038211 3.3946 0.000687
skew 0.840676 0.063438 13.2519 0.000000I wykresy
Obecnie mamy do czynienia ze spadającą zmiennością. Nie jest to przypadek, a zjawisko związane z grupowaniem wariancji. Po okresie dużej zmienności przychodzi mała (w podsumowaniu wyjaśnienie możliwej przyczyny). Z tego powodu model prognozuje spadek stopy zwrotu w lipcu, a nawet indeksu. Nie podaję wartości, bo to nie ma większego sensu - widzimy, że nie jest to prawdziwa prognoza, tylko wartość oczekiwana, średnia.
1. dla dziennych i miesięcznych stóp zwrotu WIG20 występuje efekt GARCH(1, 1). Dla ostatnich 300 dziennych lepszy jest TGARCH, dla ostatnich 300 miesięcznych EGARCH. Nie jest to przypadek. TGARCH lepiej radzi sobie z asymetrią wpływu pozytywnych i negatywnych informacji. Jak łatwo się domyślić, powinno (i jest) to bardziej widoczne dla zmian krótkoterminowych, bo wiadomości zmieniają się z dnia na dzień. Z kolei EGARCH będzie lepszy tam gdzie ujawnia się zjawisko grupowania wariancji. To bardziej tajemnicza sprawa, jak sądzę jest związana z cyklicznością przepływu kapitału. Kasa przecież generalnie spływa co miesiąc do gospodarki, a kalendarz wszyscy mają mniej więcej taki sam. Więcej kapitału to większa liczba inwestorów, to większa liczba wycen, a co za tym idzie większa rozbieżność opinii, stąd większa zmienność. Stąd oczywiste, że zjawisko grupowania wariancji prędzej będzie się odbywać w interwałach miesięcznych niż dziennych.
2. GARCH stwierdza, że zarówno dzienne, jak i miesięczne stopy zwrotu WIG20 nie zawierają autokorelacji, natomiast korelują dodatnio z ryzykiem, tj. mamy do czynienia z GARCH-in-Mean. To dobra wiadomość dla teoretyków finansów, bo po pierwsze w końcu mamy dowód, że CAPM na polskim rynku działa oraz zachowana zostaje jego efektywność (bo właśnie nie ma autokorelacji, a jest dodatni związek między ryzykiem a oczekiwaną stopą zwrotu).
3. Chociaż zdecydowanie należy odrzucić rozkład normalny, to dla log-stóp zwrotu można spokojnie przyjąć skośno-normalny.
4. Biorąc pod uwagę poprzednie punkty, należy stwierdzić że mamy do czynienia z warunkowym CAPM ze skośnością oraz efektami GARCH.
P. S. Jeżeli test Nybloma wskazuje , że model jest niestabilny i poprawianie założeń tego nie zmienia, należy skorzystać z tych testów. Alternatywą jest jeszcze CUSUMQ lub ogólniejsza rodzina testów fluktuacji (dla kwadratów stopy zwrotu), które w pewnym sensie łączą warunkowe i niewarunkowe miary.












Brak komentarzy:
Prześlij komentarz